Background.
Cryptic chromosome aberrations, i.e. those that are not observed by routine chromosome analysis (G-banding), can cause a plethora of developmental abnormalities, usually at least a combination of various dysmorphic signs and mental retardation.
Materials and methods.
We present the current molecular cytogenetic methods for detection of cryptic chromosome aberrations, with special emphasis on comparative genomic hybridisation (CGH), a DNA-based screening method.
Results.
Using comparative genomic hybridisation with high-resolution analysis, we were able to detect a chromosome aberration in 10 % of patients found to have normal karyotypes by standard chromosome analysis.
Interpretation.
Though the sensitivity of comparative genomic hybridisation is still insufficient for finding a deletion in most of the well-known microdeletion syndromes, the diagnostic yield of genomic imbalances is better than for all other laboratory investigations except routine G-banding.
Mental retardasjon (IQ < 70) forekommer hos 0,6 – 0,7 % av befolkningen (1, 2). Kromosomavvik er den vanligste kjente årsak til mental retardasjon, og ved rutinemessig kromosomanalyse gjøres det funn hos omtrent 12 % av disse personene (3). På tross av sterkt forbedret cytogenetisk, molekylærgenetisk og klinisk diagnostikk finner man hos over halvparten av mentalt retarderte barn fortsatt ikke årsaken til utviklingsavviket (4) – (6). Det er som regel god grunn til å anta at ukjente og ofte tilfeldig oppståtte gen- eller kromosomfeil likevel ligger til grunn for tilstanden. Mange av disse barna har en kombinasjon av mental retardasjon, dysmorfe trekk og misdannelser i ulike organer som passer dårlig inn i et kjent syndrombilde. Kryptiske kromosomfeil (mikrodelesjoner eller mikroduplikasjoner) kan være en mulig årsak til utviklingsavviket hos disse barna.
Standard kromosomanalyse
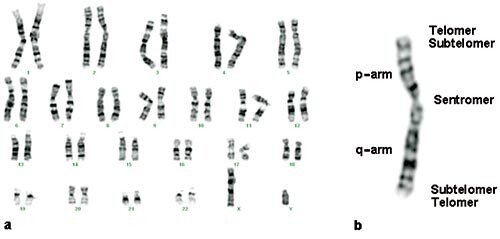
Den eneste screeningmetoden for genetisk sykdom som hittil har vært tilgjengelig, er tradisjonell karyotypering, hvor man utfører en systematisk analyse av kromosomene når de foreligger i metafase av cellesyklus (fig 1a). Kun forandringer som er større enn 5 – 15 millioner basepar (Mb), vil rutinemessig bli oppdaget, og en rekke kromosomfeil blir derfor oversett. Sensitiviteten er også avhengig av båndstrukturen – avvik ses lettere i områder med distinkte, tettsittende bånd enn i områder med diffust eller manglende båndmønster.
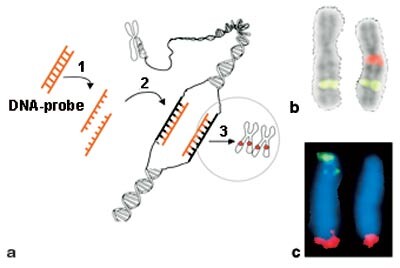
Fluorescens in situ-hybridisering (FISH)
Dersom de kliniske observasjoner gir mistanke om et kjent syndrom som skyldes en kryptisk kromosomfeil, for eksempel mikrodelesjonssyndromer som del22q11/DiGeorges syndrom, Williams syndrom, Smith-Magenis syndrom, Miller-Diekers syndrom o.a., kan dette undersøkes ved hjelp av fluorescens in situ-hybridisering (FISH) (fig 2). Det er imidlertid viktig å presisere at FISH ikke er en screeningmetode, men en undersøkelse som gjøres når kliniske symptomer og funn indikerer en delesjon (sjeldnere duplikasjon) av et bestemt kromosomområde.
Spesialundersøkelse for subtelomeriske avvik
Området innenfor telomeren (navnet på sekvensen som er lokalisert på enden av hver kromosomarm) kalles det subtelomeriske område (fig 1b). Noen subtelomeriske delesjoner gir kjente syndromer (for eksempel 4p-/Wolf-Hirschhorns syndrom, 5p-/cri du chat-syndrom, 18p- og 18q-syndrom). Delesjoner eller duplikasjoner av subtelomeriske kromosomområder er funnet hos opptil 10 % av barn med moderat til alvorlig mental retardasjon og dysmorfe trekk (7, 8). Screening for subtelomeriske avvik kan gjøres ved å anvende et subtelomerisk sett av FISH-prober (9) eller ved matrise-CGH (10). Det er større sannsynlighet for å finne subtelomeriske avvik hos et barn med mental retardasjon og dysmorfe trekk dersom barnet har annen- eller tredjegradsslektninger med liknende fenotype. Dersom man påviser et subtelomerisk avvik, vil en av foreldrene i halvparten av tilfellene ha en kryptisk balansert translokasjon mellom to kromosomender. Denne translokasjonen kan også finnes i familien for øvrig, og en familieundersøkelse (først og fremst foreldre) bør derfor utføres dersom et barn har en subtelomerisk ubalanse.
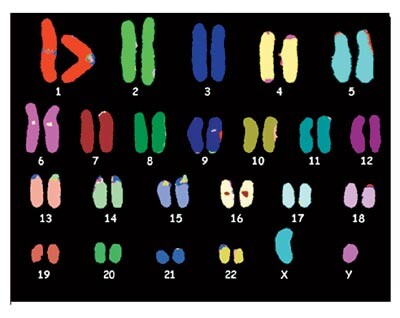
Multifarge-FISH
Som alternativ til vanlig kromosomanalyse er det utviklet screeningmetoder basert på FISH. Den ene metoden kalles SKY (spektral karyotyping) (11), den andre M-FISH (multifarge-FISH) (12). Prinsippet for begge metoder er at hvert kromosom får sin unike farge ved at kromosompreparatet hybridiseres med en blanding av ulikt fluorescerende DNA-prober for hvert kromosom. Kromosomenes fargesammensetning kan enten registreres ved bruk av filtre (M-FISH) (fig 3) eller spektral kamera (SKY). Det siste er kostbart spesialutstyr som de færreste rutinelaboratorier har tilgjengelig. Sensitiviteten er bedre enn ved vanlig kromosomanalyse dersom det foreligger utveksling av materiale mellom kromosomer (translokasjoner og insersjoner), men metoden vil som regel ikke avsløre forandringer innen samme kromosom (duplikasjoner, delesjoner og inversjoner). Metodene er særlig nyttige når det foreligger komplekse forandringer der flere kromosomer er involvert (13), og også ganske små interkromosomale forandringer kan oppdages (14). Både M-FISH og SKY er anvendt for å finne subtelomeriske kromosomavvik (13), men metodene er her lite egnet fordi sensitiviteten er for lav (15). Selv med forbedrede probesett er oppløsningen så grov at translokert materiale av en størrelse under ca. 3 Mb ikke vil kunne forventes å bli oppdaget.
DNA-markører til undersøkelse av kromosomal ubalanse
Gjennom hele genomet finnes det områder med enkle repetisjoner av baser (ofte kalt STR eller «simple tandem repeats»). Antallet repetisjoner i et bestemt område kan variere fra individ til individ og fra kromosom til kromosom – områdene er såkalt polymorfe ved at de viser naturlig lengdevariasjon i en befolkning. En genetisk markør er et slikt naturlig forekommende polymorft område med kjent kromosomlokalisering. Ved å undersøke lengden av en bestemt markør kan man skille mellom kromosomet som er arvet fra far og kromosomet som er arvet fra mor. Dersom et barn har et område på kromosomet som er deletert, vil man oppdage dette ved å sammenlikne lengden på markører i det bestemte området hos barnet med lengdene på de samme markørene hos mor og far (16). DNA-markører som er lokalisert til enden på kromosomarmene, har vært benyttet til å lete etter subtelomeriske avvik hos mentalt retarderte personer. Det ble rapportert funn hos 10 % (17).
Komparativ genomisk hybridisering (CGH)
Selv om subtelomeriske delesjoner og duplikasjoner synes å være en vesentlig årsak til mental retardasjon, kan det tenkes at små delesjoner og duplikasjoner i andre deler av kromosomene forekommer enda hyppigere. Kun et fåtall av disse vil falle inn under kjente mikrodelesjonssyndromer (se www.medgen.no for mer informasjon). Ved en CGH-analyse kan hele genomet undersøkes for slike ubalanser (fig 4). Metoden er basert på FISH-teknikken, og ble første gang beskrevet i 1992 (18). Utgangspunktet for analysen er renset DNA fra blod eller vev. Nettopp av denne grunn er CGH først og fremst blitt anvendt for å lete etter kromosomal ubalanse i solide svulster, vev som det til dels har vært vanskelig eller umulig å karyotypere (19). For analyse av medfødte kromosomavvik er CGH benyttet for nærmere karakterisering av avvik som allerede er oppdaget ved rutinemessig kromosomanalyse (20, 21). I tillegg er teknikken benyttet som en screeningmetode for subtelomeriske avvik. Man antar at CGH-analysen er sensitiv nok å oppdage de fleste subtelomeriske ubalanser (22).
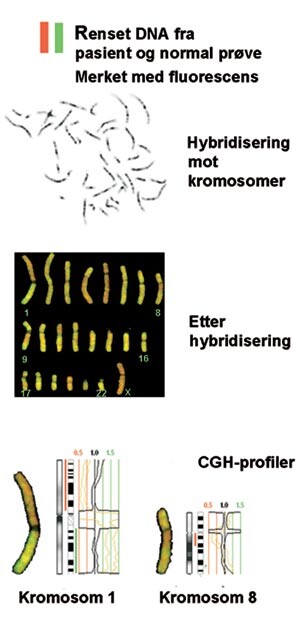
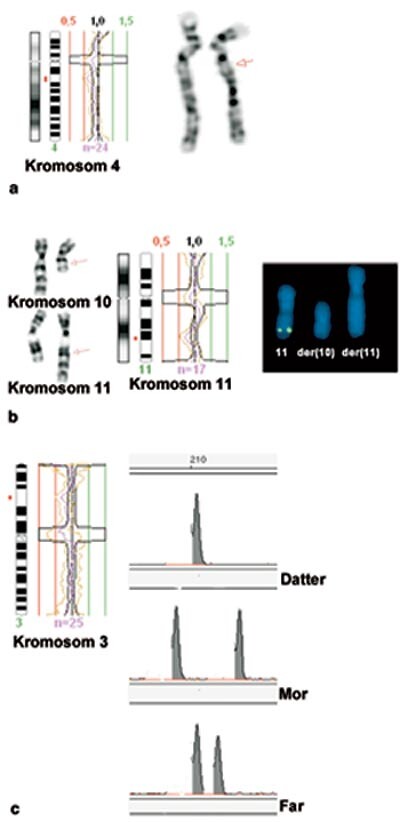
Ved å anvende analyseprogrammet høyresolusjons-CGH (HR-CGH), kan genomiske ubalanser ned til 3 Mb oppdages. Dette innebærer at sensitiviteten er 2 – 3 ganger høyere enn ved tradisjonell CGH (23). Vi har benyttet HR-CGH til diagnostikk av pasienter med mental retardasjon og dysmorfe trekk som tidligere har hatt tilsynelatende balansert kromosommønster ved vanlig kromosomanalyse (fig 5). Hittil er 116 slike barn blitt undersøkt. Hos 12 av disse har vi påvist en genomisk ubalanse. Kun i tre av tilfellene var de subtelomeriske områdene involverte. Hos ni andre forelå delesjoner eller duplikasjoner av andre deler av kromosomene. Resultatet av de 50 første undersøkte pasientene er publisert (24), og samsvarer med andre rapporterte funn (25). HR-CGH kan også benyttes til å oppdage små delesjoner ved bruddstedene ved nyoppståtte translokasjoner (fig 5b) (24, 25). Dessverre er vi fortsatt avhengig av at en kromosomal ubalanse er større enn 3 – 4 Mb for at den skal oppdages ved HR-CGH. Ved mange kjente syndromer (for eksempel Williams syndrom, del22q11-syndrom) er delesjonene under denne deteksjonsgrensen.
Screening for kromosomal ubalanse ved matrise-CGH
Screening av hele genomet for kromosomale ubalanser har nå en sensitivitet på maksimalt 3 Mb. En ytterligere bedring av sensitiviteten vil først være mulig når matrise-CGH-teknikken er ferdig utviklet. Her benyttes DNA-mikroplater med flere tusen DNA-fragmenter som representerer tilnærmet hele genomet (26, 27). Hybridiseringen av prøvene utføres på en slik DNA-matrise i stedet for på et kromosompreparat (27) – (29). Slike teknikker kan tenkes å kunne påvise ubalanser av størrelser ned til noen hundre kilobasepar (Kb), dvs. at sensitiviteten vil være omtrent ti ganger høyere enn det man kan oppnå med HR-CGH per i dag. For å komme ned i en oppløsning på 1 Mb, trengs minst 3 000 ulike DNA-fragmenter i matrisen. For å dekke hele genomet må matrisen inneholde minst 35 000 fragmenter av en størrelse på 150 – 200 Kb. Arbeidet med å plukke ut fragmenter og å verifisere posisjonene til disse er svært tidkrevende. I tillegg kommer de tekniske utfordringene som selve matriseteknologien innebærer. Det er blitt fremstilt spesial-CGH-matriser med DNA-fragmenter svarende til begrensede deler av genomet, for eksempel de subtelomeriske områdene (10). Helgenomiske matriser finnes, men av tekniske og kostnadsmessige årsaker er disse kun blitt anvendt i liten utstrekning. For høy sensitivitet kan paradoksalt nok bli det største problemet i fremtiden. Mye tyder på at det humane genom er temmelig ustabilt. Før man får samlet nok erfaring, vil tolkingen av resultatene være vanskelig fordi man ikke kjenner hvilke områder som har naturlig forekommende polymorfisme for delesjoner, duplikasjoner og amplifikasjoner (30).
Konklusjon
Kryptiske og vanligvis tilfeldig oppståtte kromosomavvik er en meget hyppig forekommende årsak til medfødte utviklingsforstyrrelser med mental retardasjon. Genomisk ubalanse forårsaket av slike avvik kan detekteres med komparativ genomisk hybridisering dersom delesjonen eller duplikasjonen er over 3 – 4 Mb. Den store fordelen med CGH er at alle deler av kromosomene blir undersøkt og at man kun trenger DNA fra pasienten til slik analyse. Når man i fremtiden kan anvende mer sensitiv matrise-CGH for å lete etter slike avvik, forventes det at vår evne til å finne årsaken til medfødte utviklingsavvik forbundet med mental retardasjon vil bli betydelig forbedret.
Kryptiske kromosomavvik er vanskelige eller umulige å oppdage ved rutinemessig kromosomanalyse
Slike avvik er en viktig, kanskje den viktigste, årsak til medfødte utviklingsavvik med mental retardasjon
Komparativ genomisk hybridisering er den eneste generelle screeningmetode for påvisning av genomisk ubalanse som følge av kryptiske kromosomavvik
Med CGH-metoden kan vi i dag påvise et kryptisk kromosomavvik hos 10 % av mentalt retarderte og dysmorfe barn med tilsynelatende normal karyotype
- 1.
Roeleveld N, Zielhuis GA, Gabreels F. The prevalence of mental retardation: a critical review of recent literature. Dev Med Child Neurol 1997; 39: 125 – 32.
- 2.
Strømme P. Epidemiological, genetic and neurological aspects of mental retardation. A Norwegian population-based study of children born between 1980 and 1985. Doktoravhandling. Oslo: Universitetet i Oslo, 2000.
- 3.
Curry CJ, Stevenson RE, Aughton D, Byrne J, Carey JC, Cassidy S et al. Evaluation of mental retardation: recommendations of a consensus conference. Am J Med Genet 1997; 72: 468 – 77.
- 4.
Flint J, Wilkie AO. The genetics of mental retardation. Br Med Bull 1996; 52: 453 – 64.
- 5.
Ørstavik KH. Psykisk utviklingshemning. Tidsskr Nor Lægeforen 1997; 117: 1438.
- 6.
Strømme P. Syndromer – riktig diagnose gir bedre oppfølging. Tidsskr Nor Lægeforen 1998; 118: 1539.
- 7.
Knight SJ, Flint J. Perfect endings: a review of subtelomeric probes and their use in clinical diagnosis. J Med Genet 2000; 37: 401 – 9.
- 8.
de Vries BBA, White SM, Knight SJL, Regan R, Homfray T, Young ID et al. Clinical studies on submicroscopic subtelomeric rearrangements: a checklist. J Med Genet 2001; 38: 145 – 50.
- 9.
Brown J, Saracoglu K, Uhrig S, Speicher MR, Eils R, Kearney L. Subtelomeric chromosome rearrangements are detected using an innovative 12-color FISH assay (M-TEL). Nat Med 2001; 7: 497 – 501.
- 10.
Veltman JA, Schoenmakers EFPM, Eussen BH, Janssen I, Merkx G, van Cleef B et al. High-throughput analysis of subtelomeric chromosome rearrangements by use of array-based comparative genomic hybridization. Am J Hum Genet 2002; 70: 1269 – 76.
- 11.
Schröck E, du Manoir S, Veldman T, Schoell B, Wienberg J, Ferguson-Smith MA et al. Multicolor spectral karyotyping of human chromosomes. Science 1996; 273: 494 – 7.
- 12.
Speicher MR, Ballard SG, Ward DC. Karyotyping human chromosomes by combinatorial multi-fluor FISH. Nat Genet 1996; 12: 368 – 75.
- 13.
Uhrig S, Schuffenhauer S, Fauth C, Wirtz A, Daumer-Haas C, Apacik C et al. Multiplex-FISH for pre- and postnatal diagnostic applications. Am J Hum Genet 1999; 65: 448 – 62.
- 14.
Bezrookove V, Hansson K, van der Burg M, van der Smagt JJ, Hilhorst-Hofstee Y, Wiegant J et al. Individuals with abnormal phenotype and normal G-banding karyotype: improvement and limitations in the diagnosis by the use of 24-colour FISH. Hum Genet 2000; 106: 392 – 8.
- 15.
Lee C, Gisselsson D, Jin C, Nordgren A, Ferguson DO, Blennow E et al. Limitations of chromosome classification by multicolor karyotyping. Am J Hum Genet 2001; 68: 1043 – 7.
- 16.
Rosenberg MJ, Vaske D, Killoran CE, Ning Y, Wargowski D, Hudgins L et al. Detection of chromosomal aberrations by a whole-genome microsatellite screen. Am J Hum Genet 2000; 66: 419 – 27.
- 17.
Rio M, Molinari F, Heuertz S, Ozilou C, Gosset P, Raoul O et al. Automated fluorescent genotyping detects 10 % of cryptic subtelomeric rearrangements in idiopathic syndromic mental retardation. J Med Genet 2002; 39: 266 – 70.
- 18.
Kallioniemi OP, Kallioniemi A, Piper J, Isola J, Waldman FM, Gray JW et al. Optimizing comparative genomic hybridization for analysis of DNA sequence copy number changes in solid tumors. Genes Chromosomes Cancer 1994; 10: 231 – 43.
- 19.
Gray JW, Collins C. Genome changes and gene expression in human solid tumors. Carcinogenesis 2000; 21: 443 – 52.
- 20.
Breen CJ, Barton L, Carey A, Dunlop A, Glancy M, Hall K et al. Applications of comparative genomic hybridisation in constitutional chromosome studies. J Med Genet 1999; 36: 511 – 7.
- 21.
Rigola MA, Carrera M, Ribas I, De La Iglesia C, Mendez B, Egozcue J et al. Identification of two de novo partial trisomies by comparative genomic hybridization. Clin Genet 2001; 59: 106 – 10.
- 22.
Joly G, Lapierre J-M, Ozilou C, Gosset P, Aurias A, de Blois M-C et al. Comparative genomic hybridisation in mentally retarded patients with dysmorphic features and a normal karyotype. Clin Genet 2001; 60: 212 – 9.
- 23.
Kirchhoff M, Gerdes T, Rose H, Maahr J, Ottesen AM, Lundsteen C. Detection of chromosomal gains and losses in comparative genomic hybridization analysis based on standard reference intervals. Cytometry 1998; 31: 163 – 73.
- 24.
Ness GO, Lybæk H, Houge G. Usefulness of high-resolution comparative genomic hybridization (CGH) for detecting and characterizing constitutional chromosome abnormalities. Am J Med Genet 2002; 113: 125 – 36.
- 25.
Kirchhoff M, Rose H, Lundsteen C. High resolution comparative genomic hybridisation in clinical cytogenetics. J Med Genet 2001; 38: 740 – 4.
- 26.
Antonarakis SE. BACking up the promises. Nat Genet 2001; 27: 230 – 2.
- 27.
Snijders AM, Nowak N, Segraves R, Blackwood S, Brown N, Conroy J et al. Assembly of microarrays for genomewide measurement of DNA copy number. Nat Genet 2001; 29: 263 – 4.
- 28.
Solinas-Toldo S, Lampel S, Stilgenbauer S, Nickolenko J, Benner A, Dohner H et al. Matrix-based comparative genomic hybridization: biochips to screen for genomic imbalances. Genes Chromosomes Cancer 1997; 20: 399 – 407.
- 29.
Pinkel D, Segraves R, Sudar D, Clark S, Poole I, Kowbel D et al. High resolution analysis of DNA copy number variation using comparative genomic hybridization to microarrays. Nat Genet 1998; 20: 207 – 11.
- 30.
Stankiewicz P, Lupski JR. Molecular-evolutionary mechanisms for genomic disorders. Curr Opin Genet Dev 2002; 12: 312 – 9.